Practice initiation cluster i-Trop
| Description | Exercices pour cluster i-Trop |
|---|---|
| Cours lié | HPC |
| Auteur | Ndomassi TANDO (ndomassi.tando@ird.fr) |
| Date de création | 25/11/2019 |
| Dernière date de modification | 08/09/2023 |
Sommaire
- Preambule: Logiciel à installer pour se connecter à un serveur Linux distant
- Practice 1: Se connecter à un serveur Linux via
ssh - Practice 2: Réserver un coeur d'un noeud en intéractif et créer son répertoire de travail
- Practice 3: Transférer des fichiers avec filezilla et installer vos clés ssh
sftp - Practice 4: Transférer des données vers les noeuds
scp - Practice 5: Utiliser module environment pour charger ses outils
- Practice 6: Lancer des analyses
- Practice 7: Transférer des données vers le serveur san
scp - Practice 8: Supprimer son répertoire temporaire
- Practice 9: Lancer un job via sbatch
- Lien
- Licence
Préambule
Se connecter à un serveur Linux depuis Windows
| Platforme | Logiciel | Description | url |
|---|---|---|---|
 |
mobaXterm | Un terminal avancé pour Windows avec serveur X11 server | Télécharger |
 |
putty | Putty permet de se connecter à un serveur Linux depuis une machine Windows | Télécharger |
Transférer et copier des fichiers depuis votre ordinateur vers des serveurs Linux avec SFTP (SSH File Transfer Protocol)
| Platforme | Logiciel | Description | url |
|---|---|---|---|
   |
 filezilla filezilla |
Client FTP et SFTP | Télécharger |
Visualiser et éditer des fichiers depuis son ordinateur avant de le transférer vers un serveur Linux distant ou directement depuis le serveur distant
| Type | Logiciel | url |
|---|---|---|
| Distant, mode console | nano | Tutoriel |
| Distant, mode console | vi | Tutoriel |
| Distant, mode graphique | komodo edit | Télécharger |
| Editeur Linux & Windows | Notepad++ | Télécharger |
Practice 1: Se connecter à un serveur Linux via ssh
Avec mobaXterm:
- Cliquer sur le bouton session, puis cliquer sur SSH.
- Dans le champ "remote host" , taper: bioinfo-master1.ird.fr
- Cocher la case "specify username" et entrer votre login
- Dans la console, entrer le mot de passe quand demandé.
Attention pour des raisons de sécurité, le mot de passe n'apparait pas à l'écran - Taper la commande
sinfoet commenter le resultat - Taper la commande
sinfo -N nodes --longet noter les informations supplémentaires - Taper la commande
scontrol show nodes
Practice 2: Réserver un coeur d'un noeud en intéractif et créer son répertoire de travail
- Taper la commande
squeueet noter le résultat - Taper la commande
squeue -u your_loginavec "your_login" à remplacer avec votre login - Plus de details avec la commande:
squeue -O "username,name:40,partition,nodelist,NumCPUs,state,timeused,timelimit" - Taper la commande
srun -p short --pty bash -ipuissqueueencore - Créer votre propre répertoire dans le /scratch du noeud:
cd /scratch
mkdir login
avec login : le nom de votre choix
Practice 3 : Transférer des fichiers avec filezilla et installer ses clés ssh sftp
Télécharger et installer FileZilla
Ouvrir FileZilla et sauvegarder IRD cluster dans "site manager"
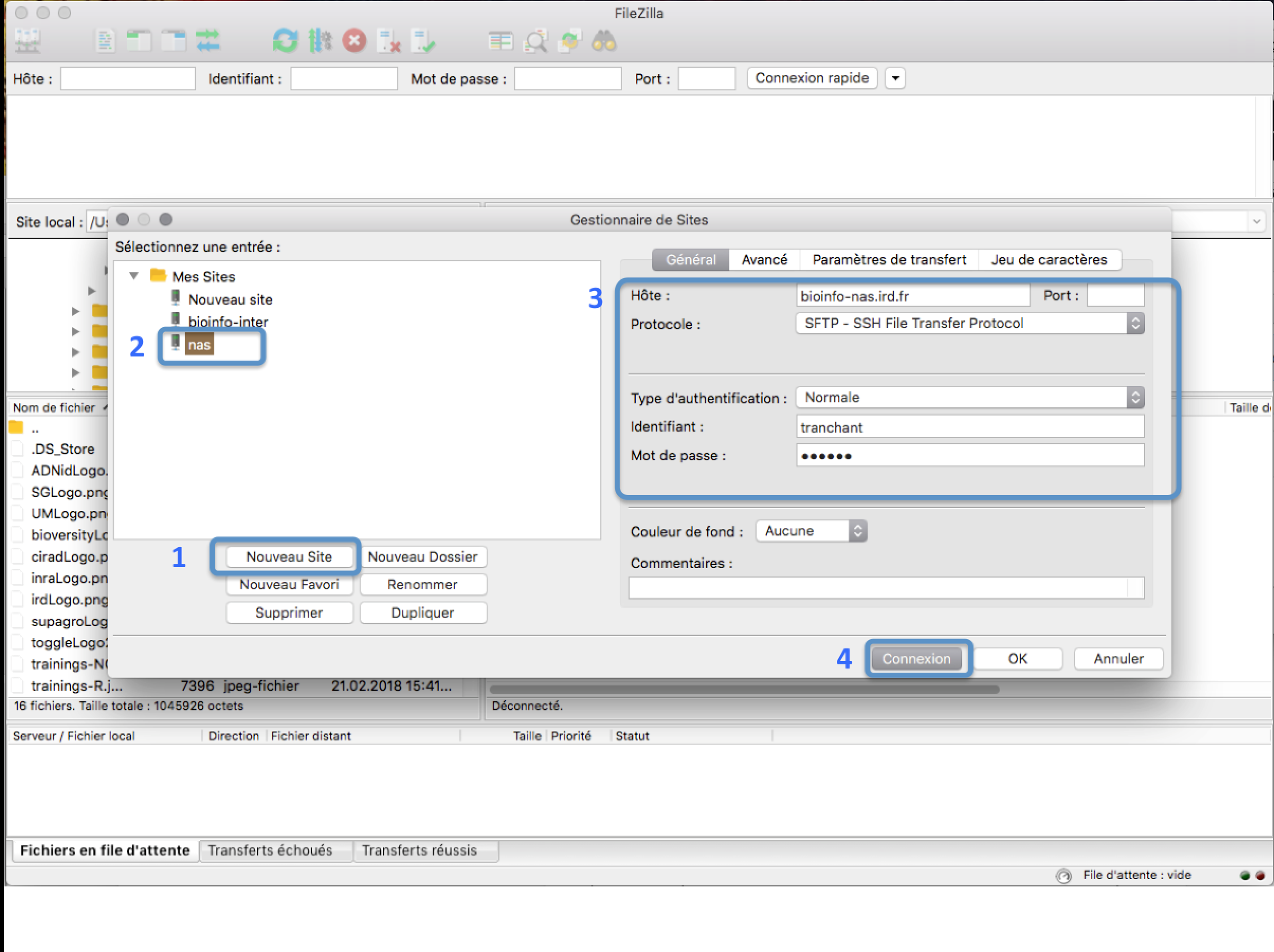
Dans le menu FileZilla, aller dans File > Site Manager. Puis suivre ces étapes:
- Cliquer New Site.
- Ajouter un nom personnalisé pour ce nouveau site.
- Ajouter bioinfo-san.ird.fr pour avoir accès à votre /home/login
- Mettre Logon Type à "Normal" et mettre ses identifiants de connexion au cluster
- Presser le bouton the "Connect".
Transfert de fichiers
- Depuis votre ordinateur vers le cluster : faire un glisser-déposer d'un fichier depuis la colonne de gauche vers celle de droite
- Depuis le cluster vers votre ordinateur : faire un glisser-déposer d'un fichier depuis la colonne de droite vers celle de gauche
- Récupérer le fichier ssh-login.zip (avec login votre compte cluster) depuis la fenêtre de droite dans le répertoire /home/login
Installation des clés ssh sur votre ordinateur
Suivez les instructions ici en fonction de votre OS:
https://bioinfo.ird.fr/index.php/tutorials-howto-add-ssh-keys-to-your-computer-copy/
Practice 4: Transférer des données vers les noeuds
- En utilisant scp, transférer le répertoire
Ebolasitué dans/share/formation/Slurm/TPassemblydans votre répertoire de travail - Vérifier le résultat avec la commande ls
Practice 5: Utiliser module environment pour charger ses outils
- Charger le module abyss 2.2.5
- Vérifier si le module est chargé
Practice 6 : Lancer des analyses
Effectuer un assemblage avec abyss-pe
Avec abyss , nous réassemblons les séquences en utilisant les fichiers fastq ebola1.fastq et ebola2.fastq
Lancer les commandes
module load abyss/2.2.5abyss-pe k=35 in='ebola1.fastq ebola2.fastq' name=k35Practice 7: Transférer des données vers le serveur san
- En utilisant scp, transférer les résultats de
/scratch/loginvers votre/home/login - Vérifier si le transfert est OK avec ls
Practice 8: Supprimer son répertoire temporaire
cd /scratch
rm -r loginexitPractice 9: Lancer un job via sbatch
En suivant les différentes étapes effectuées jusqu'ici, créer un script our lancer les analyses du practice6:
1ere étape: créer une section Slurm dans votre script
1) Donner un nom à son job
2) Préciser son adresse email
3) Choisir la partition short
2eme étape : taper les commandes que vous voulez que le script lance:
1) créer un répertoire personnel dans /scratch avec mkdir
2) En utilisant scp, transférer le répertoire the Ebola situé dans /share/formation/Slurm/TPassembly dans votre répertoire de travail
3) Charger abyss version 2.2.5 avec module environment
4) Dans le répertoire Ebola, lancer la commande suivante:
abyss-pe k=35 in='ebola1.fastq ebola2.fastq' name=k355) En utilisant scp, transférer ses results depuis le /scratch/login vers son /home/login
6) Suppression du répertoire personnel de /scratch
Lancer les commandes suivantes pour avoir des infos sur le job terminé :
seff <JOB_ID>
sacct --format=JobID,elapsed,ncpus,ntasks,state,node -j <JOB_ID>Bonus:
lancement d'une analyse en 4 étapes:
1) Effectuer un alignement multiple avec l'outil nucmer
2) Filtrer ces alignments avec l'outil delta-filter
3) Générer un tableau facile à parser avec l'outil show-coords
4) Générer une image png avec mummerplot
-
Récupérer le script /projects/medium/formation/Slurm/scripts/alignment_slurm.sh dans son /home/login
-
modifier la section Slurm section et les variables
-
lancer le script avec sbatch:
sbatch alignment.sh-
Taper
ls -altrdans votre/home/login. Que constatez-vous? -
Lancer les commandes suivantes pour obtenir des infos sur le job terminé:
seff <JOB_ID>
sacct --format=JobID,elapsed,ncpus,ntasks,state,node -j <JOB_ID>-
Ouvrir filezilla et récupérer l'image png sur votre ordinateur
-
Lancer la commande suivante pour nettoyer le /scratch du noeud
sh /opt/scripts/scratch-scripts/scratch_use.shet choisir le numéro de noeud utilisé
Liens
- Cours liés : Linux for Dummies
- Tutoriaux : Linux Command-Line Cheat Sheet







